Медиатека
Рибосомный профайлинг: изучение трансляции на полногеномном уровне
Оглавление:
•
Описание метода
•
Экспериментальный протокол
•
Модификации метода
•
Примеры работ с применением рибосомного профайлинга
Трансляция является фундаментальным, жизненно необходимым клеточным процессом и одним из этапов реализации генетической информации, в ходе которого осуществляется синтез белков на основе закодированной в мРНК последовательности. Трансляция – сложный многостадийный процесс, в котором задействовано много «участников»: факторы трансляции, аминокислоты, тРНК, регуляторные элементы и т.д. Ключевым функциональным участником процесса трансляции являются рибосомы – высокомолекулярные рибонуклеопротеидные комплексы со сложной структурой и организацией, которые осуществляют декодирование заключенной в мРНК информации и непосредственно построение полипептидной цепи. Центральная роль, которую играет трансляция в клетке, способствовала разработке экспериментальных подходов к анализу белков, продуцируемых клеткой (протеома), и процесса их синтеза. В нашей статье мы расскажем о революционном методе в области изучения биосинтеза белка – рибосомном профайлинге, который позволяет изучать трансляцию мРНК и ассоциированные с ней котрансляционные процессы на полногеномном уровне.
Описание метода
Рибосомный профайлинг (РП) – метод идентификации транслируемых в клетке мРНК (транслятома), основанный на глубоком секвенировании защищенных рибосомами фрагментов мРНК. Метод позволяет проводить глобальный анализ трансляции в клетках, выявляя положения полного набора транслирующих рибосом. Метод был разработан в 2009 году Николасом Инголиа в лаборатории Джонатана Вайсмана в Калифорнийском Университете и впервые применен к клеткам дрожжей 1, а затем успешно опробован на эмбриональных стволовых клетках мышей. 2 До этого момента не существовало простого высокопроизводительного метода измерения экспрессии генов на уровне трансляции, хотя был накоплен уже большой объем статичной информации о составе клетки благодаря масс-спектрометрии (о составе протеома) и секвенированию РНК (о составе транскриптома). Предоставляя полногеномную информацию о синтезе белка, рибосомный профайлинг заполнил технологический пробел, существующий между нашими возможностями к количественной оценке транскриптома и протеома.
Короткие фрагменты мРНК длиной примерно 30 нуклеотидов (нт), которые физически окружены рибосомой и защищены от расщепления нуклеазами, называют рибосомными футпринтами. Такие футпринты преобразуются в библиотеку фрагментов ДНК и анализируются с помощью секвенирования нового поколения. Каждый секвенированный футпринт идентифицирует положение одной рибосомы, показывая, какой транскрипт транслировался этой рибосомой и в каком месте кодирующей последовательности она была «захвачена» во время лизиса клетки. Современные технологии глубокого секвенирования позволяют анализировать сотни миллионов отдельных коротких «чтений» (ридов) в одном эксперименте. Применительно к библиотекам рибосомных футпринтов это дает исчерпывающее представление о процессе трансляции и позволяет ответить на многие фундаментальные вопросы, в том числе, какие мРНК транслируются в данный момент в конкретном типе клеток в определенных условиях, какие мРНК транслируются активнее, как рибосомы распределены вдоль транскрипта в процессе трансляции, как происходит регуляция трансляции при изменении условий среды и т.д. РП применяется для решения удивительно широкого спектра механистических и физиологических вопросов, а полученные данные обеспечивают всестороннее представление о жизнедеятельности клеток. 1, 3
Экспериментальный протокол
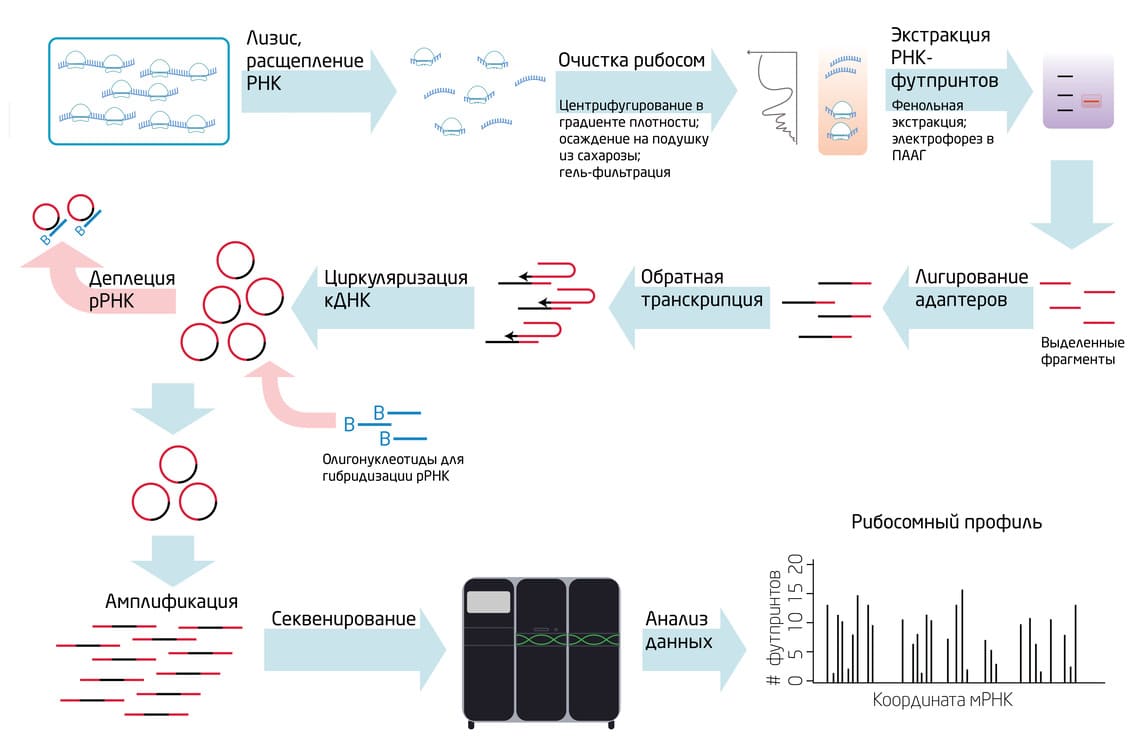
Ниже мы разберем основные этапы экспериментального протокола РП. 4
Рис. 1. Основные этапы протокола рибосомного профайлинга. 5
1. Остановка рибосом и лизис клеток.
Для того чтобы зафиксировать рибосомы на мРНК, обычно применяют антибиотики, ингибирующие трансляцию (например, циклогексимид). Затем, в зависимости от типа клеток или тканей, выбирается наиболее подходящий способ лизиса.
2. Обработка лизата эндонуклеазами рестрикции (РНКазами).
В результате расщепляются свободные РНК, а также мРНК в составе полисом, высвобождая отдельные моносомы. Защищенными от деградации остаются фрагменты мРНК внутри рибосом – футпринты.
3. Очистка рибосом и экстракция РНК-футпринтов.
После расщепления нуклеазой происходит очистка комплектов 80S рибосом с футпринтами от остальных компонентов лизата. Основные способы очистки рибосом: центрифугирование в градиенте плотности сахарозы (самый распространенный способ); осаждение через «подушку» из 1М сахарозы; гель-фильтрация рибосом; в некоторых системах можно использовать генетические манипуляции для добавления эпитопных меток к рибосомам, что позволяет проводить аффинную очистку. Затем РНК выделяют из моносом методом фенольной экстракции и разделяют электрофорезом в акриламидном геле РНК-фрагменты такой длины, которая соответствует длине фрагмента мРНК, защищенного рибосомой (28-34 нт).
4. Лигирование адаптеров и обратная транскрипция.
К полученным фрагментам добавляют последовательности адаптеров, необходимых для последующих этапов и секвенирования. Затем проводят реакцию обратной транскрипции для конвертации РНК в ДНК.
5. Циркуляризация кДНК и деплеция рибосомной кДНК.
Концы фрагментов кДНК лигируют друг на друга с помощью фермента цирклигазы с образованием кольцевых фрагментов. Загрязнение образцов рибосомной РНК, которое случается из-за расщепления интактных рибосом, существенно снижает количество информативных данных. Поэтому проводят реакцию деплеции рибосомной кДНК путем гибридизации биотинилированных олигонуклеотидов и аффинной хроматографии на стрептавидиновых носителях.
6. Амплификация, секвенирование и анализ данных.
Очищенные фрагменты кДНК амплифицируют с помощью полимеразной цепной реакции (ПЦР) и определяют их нуклеотидные последовательности методами массового параллельного секвенирования (методы второго поколения). Дальнейшая обработка полученных данных секвенирования осуществляется с помощью биоинформатического анализа. Картирование ридов обычно осуществляется на данные секвенирования тотальной мРНК, образцы для которого отбирают на стадии лизиса и готовят параллельно с образцами для рибосомного профайлинга.
Модификации метода
Одна из первых модификаций метода заключается в использовании различных ингибиторов трансляции, действующих на рибосому. Так, некоторые ингибиторы приводят к обогащению рибосомами стартовых кодонов мРНК, другие способны останавливать рибосомы в различных конформациях, что позволяет анализировать конформационные распределения состояний рибосом в различных условиях. 6
Следующий подход позволил изучать процесс сканирования – движение 43S преинициаторного комплекса вдоль 5`-нетранслируемой области (5`-НТО, или лидерная последовательность) мРНК в поисках стартового кодона. Главным препятствием в изучении стадии сканирования является то, что 43S комплекс и сканирующие комплексы являются по своей природе нестабильными и разрушаются в процессе выделения из клеток при стандартном протоколе. Для решения данной проблемы был разработан метод фиксации трансляционных комплексов в клетках за счет химического сшивания с помощью формальдегида. 6, 7
Эволюция технологии РП привела к появлению его особой модификации, названной селективным рибосомным профайлингом 8. Суть модификации заключается в выделении после обработки рибонуклеазой части рибосом, ассоциированных с определенным белком. Это достигается путем выделения рибосомных комплексов либо с помощью антител к интересующему белку, либо с помощью тэга, введенного в этот белок, после чего из отобранных комплексов выделяют фрагменты мРНК и секвенируют их. Селективный РП применяют также для исследования котрансляционной сборки мультисубъединичных белковых комплексов и, в частности, функций котрансляционных шаперонов. 6
В еще одной модификации РП 9 предлагается анализировать фракции футпринтов разных размеров, выходящие за пределы 25-35 нт при стандартном протоколе. Это позволяет изучать «дисомы» и столкновения более двух рибосом. При сближении двух рибосом участок мРНК между ними остается защищен и имеет более протяженную длину – 40-80 нт. Данные рибосомного профайлинга позволяют изучить причины и механизмы таких событий. 6
Примеры работ с применением рибосомного профайлинга
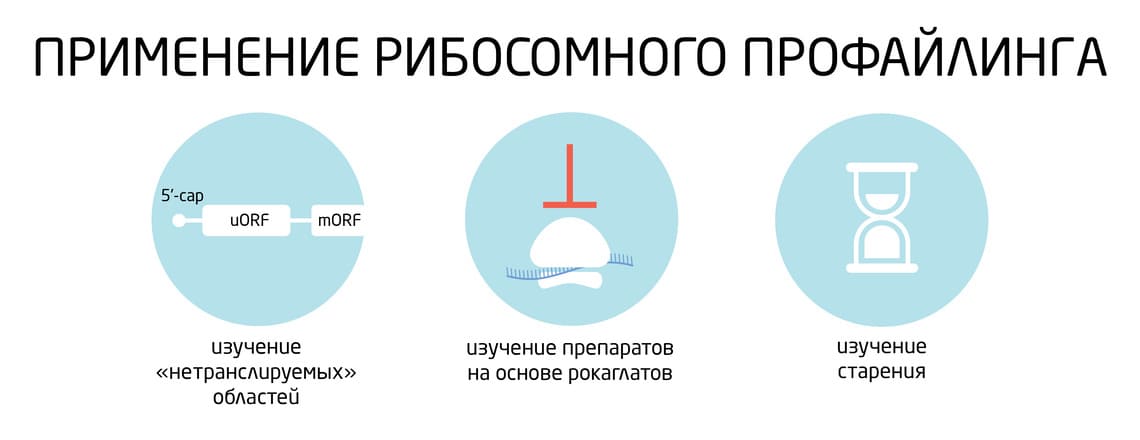
Преобладание футпринтов рибосом в лидерных последовательностях мРНК – одна из самых поразительных особенностей, которые были обнаружены в первых экспериментах с применением РП 1, 2. Это значит, что «нетранслируемые» области оказались очень даже транслируемыми. Такие последовательности в 5`НТО, названные uORF (от англ. upstream open reading frame), обладают мощным регуляторным потенциалом – способны как ингибировать трансляцию основной рамки считывания, так и стимулировать этот процесс, а потому привлекают особое внимание исследователей и активно изучаются.
Более того, многие uORF начинаются не с классического стартового кодона AUG, а с неканонических, например, CUG. Синтез пептидов с альтернативных стартовых кодонов индуцируется в условиях ингибирования ключевого инициаторного фактора eIF2 и зависит от пока плохо изученного фактора eIF2A. В одной из работ методом рибосомного профайлинга на модели мышиной SOX2-опосредованной плоскоклеточной карциномы был выявлен сдвиг в сторону eIF2A-зависимой инициации трансляции с альтернативных кодонов. 10 Согласно этим данным происходила стимуляция трансляции транскриптов многих онкогенов, прогрессирование опухоли зависело от фактора eIF2A, что указывает на причинно-следственную связь между неканонической трансляцией в 5'-НТО и раком.
С помощью РП был изучен механизм действия противоопухолевых препаратов на основе рокаглатов, нацеленных на аппарат трансляции. 11 Рокаглаты представляют собой класс природных соединений, мишенью которых является эукариотический фактор инициации eIF4A. Было показано, что рокаглаты ингибируют трансляцию специфических мРНК, предполагая, что такое целенаправленное ингибирование может объяснить их селективность в отношении раковых клеток. Путем измерения относительной чувствительности различных транскриптов к лечению рокаглатом, которая сильно отличалась от их чувствительности к гиппуристанолу, более традиционному ингибитору eIF4A, рибосомный профайлинг дополнительно прояснил уникальный репрессивный механизм этих препаратов. Вместо того чтобы имитировать потерю функции eIF4A, препараты рокаглата застопаривают eIF4A на определенных последовательностях РНК, обогащенных пуриновыми основаниями, что в результате служит препятствием для инициации трансляции. Таким образом, транскрипты с такими лидерами особенно чувствительны к обработке рокаглатом.
Рибосомный профайлинг задействован на передовой в изучении многих острых научных, клинических и социальных проблем. Еще один пример – попытка изучить процесс старения и его молекулярные механизмы на уровне трансляции 12. Изменение синтеза белка с возрастом было спорным вопросом в течение некоторого времени. Хотя считается, что общий синтез белка у млекопитающих с возрастом снижается, детали механизма остаются неясными. В одной из работ исследователи применили РП и секвенирование РНК для анализа возрастных изменений в печени и почках мышей на протяжении жизни. Новые транскриптомные данные коррелируют с ранее полученными результатами и указывают на тот факт, что возрастные изменения в экспрессии генов проявляются преимущественно на транскрипционном уровне. Самые значимые изменения с возрастом наблюдались в экспрессии генов, ассоциированных с такими процессами, как воспаление, регуляция артериального давления, биосинтез липидов и глюкокортикоидов, деградация протеасомных белков, митохондриальная активность и окислительный стресс. Рибосомный профайлинг в свою очередь позволил охарактеризовать функциональные группы генов, экспрессия которых с возрастом изменяется как на уровне транскрипции, так и на уровне трансляции. К ним относятся транскрипты, кодирующие митохондриальные белки, фактор транскрипции Irf7, связанный с иммунитетом, клинически значимые факторы гемостаза F13a1 и Vwf, связанная с ферроптозом пероксидаза Gpx4 и две субъединицы ферритина, Fth1 и Ftl1. В работе также были выявлены гены-кандидаты, которые могут служить маркерами старения в исследованных тканях. Кроме того, ученые идентифицировали специфическую трансляционную дерегуляцию по мере старения для ряда транскриптов, кодирующих компоненты клеточного аппарата синтеза белка. Еще одно интересное наблюдение описывает различия в паттернах распределения рибосом вдоль мРНК у молодых и старых животных, отражая дополнительный уровень возрастных изменений в трансляции по мере старения животных. 12
Подходы, основанные на рибосомном профайлинге, произвели революцию в изучении каждого этапа синтеза белка in vivo. Разнообразный, в том числе количественный характер данных РП предоставляет беспрецедентную возможность исследовать и моделировать сложные клеточные процессы, а улучшенные экспериментальные протоколы в сочетании с другими аналитическими методами в будущем обеспечат более глубокое понимание регулирующих трансляцию факторов и её влияния на функцию белков и физиологию клеток.
Литература
1. Ingolia N.T. et al. Genome-wide analysis in vivo of translation with nucleotide resolution using ribosome profiling. Science, 2009.
2. Ingolia N.T., Lareau L.F., Weissman J.S. Ribosome profiling of mouse embryonic stem cells reveals the complexity and dynamics of mammalian proteomes. Cell, 2011.
3. Ingolia N.T., Hussmann J.A., Weissman J.S. Ribosome Profiling: Global Views of Translation. Cold Spring Harbor Perspectives in Biology, 2019.
4. Ingolia N.T. et al. The ribosome profiling strategy for monitoring translation in vivo by deep sequencing of ribosome-protected mRNA fragments. Nature Protocols, 2012.
5. Michel A.M., Baranov P.V. Ribosome profiling: a Hi-Def monitor for protein synthesis at the genome-wide scale. Wiley Interdisciplinary Reviews: RNA, 2013.
6. Андреев Д.Е., Смирнова В.В., Шатский И.Н. Модификации рибосомного профайлинга, позволяющие получить новые данные о регуляции трансляции. Биохимия, 2021.
7. Archer S.K. et al. Dynamics of ribosome scanning and recycling revealed by translation complex profiling. Nature, 2016.
8. Oh E. et al. Selective ribosome profiling reveals the cotranslational chaperone action of trigger factor in vivo. Cell, 2011.
9. Guydosh N.R., Green R. Dom34 rescues ribosomes in 3' untranslated regions. Cell, 2014.
10. Sendoel A. et al. Translation from unconventional 5' start sites drives tumour initiation. Nature, 2017.
11. Iwasaki S., Floor S.N., Ingolia N.T. Rocaglates convert DEAD-box protein eIF4A into a sequence-selective translational repressor. Nature, 2016.
12. Anisimova A.S. et al. Multifaceted deregulation of gene expression and protein synthesis with age. Proceedings of the National Academy of Sciences of the USA, 2020.




.jpg)