- Главная страница
- Медиатека
- Информационные статьи
- От практики к теории: новая математическая модель репликации E. Coli: поддержка компании «Хеликон»
Медиатека
От практики к теории: новая математическая модель репликации E. Coli
Данные электронной микроскопии, накопленные за последние десятилетия, позволили ученым значительно продвинуться в понимании молекулярных и генетических механизмов жизнедеятельности микроорганизмов. Однако сегодня инструментальная наука уступает методам «современности» в некоторых областях. Так, с помощью компьютерного и математического моделирования авторам работы, опубликованной в Nature Communications, удалось «подглядеть» за процессом, который все еще невозможно «разглядеть» под микроскопом, – инициацией репликации в бактериальной клетке 1.
Модельным объектом исследования стала кишечная палочка Escherichia coli (Е. coli). Инициация репликации ДНК у этой бактерии точно синхронизирована с жизнедеятельностью клетки, а значит, зависит от скорости роста и клеточного деления (а они, в свою очередь, – от условий окружающей среды). В каждой точке начала репликации синтез бактериальной ДНК запускается только один раз за клеточный цикл, синхронно во всех сайтах начала репликации. При этом репликация начинается только тогда, когда масса клетки достигает определенного значения, так называемой «инициаторной массы» 2, которая рассчитывается на каждую область начала репликации.
Согласно одной из экспериментальных моделей, модели «переключения инициатора», этот процесс регулируется белком DnaA, а именно уровнем его активации и пороговой концентрации 1, 3. В течение клеточного цикла концентрация белка в клетке увеличивается пропорционально её объему, а репликация запускается только тогда, когда количество DnaA достигает пороговых значений. Контроль действия белка на репликацию описывается в модели «титрования». В этом случае пороговая концентрация белков, необходимая для запуска репликации, зависит от количества «сайтов титрования» – функциональных участков ДНК, находящихся в точках начала репликации хромосомы (oriC), или DnaA-боксов 4, 5. Только когда все DnaA-боксы оказываются связанными с белком-инициатором и его концентрация превышает число всех внутриклеточных DnaA-боксов, начинается синтез ДНК. После освобождения первой точки начала репликации от DnaA повышение его уровня в клетке запускает новый раунд репликации на других доступных сайтах.
С помощью симуляции клеточного деления учёные создали единую модель контроля инициации в клетках E. coli 1. Оказалось, что в неблагоприятных условиях окружающей среды, т.е. при низких скоростях роста клеток, механизм титрования способствует стабилизации клеточного цикла, повышая стабильность переключения между сайтами. При более высоких скоростях роста титрование было менее эффективным и часто приводило к преждевременной повторной инициации. В этом случае запускался механизм накопления инициатора (Рис. 1B), контролирующий баланс активации и дезактивации белка. Этот механизм позволяет поддерживать устойчивые ритмы клеточного деления при высоких скоростях роста, наблюдаемых при благоприятных условиях среды.
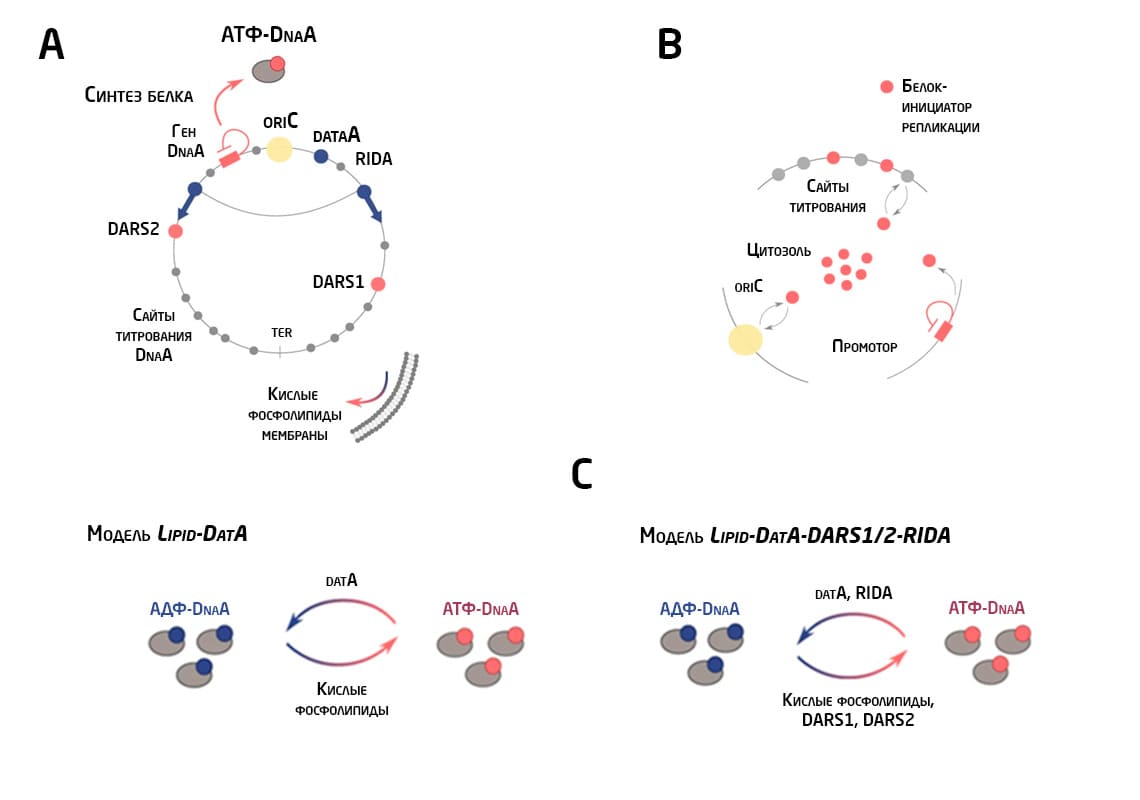
Рисунок 1. A. Схематическое изображение процесса репликации в хромосоме E. coli: начало репликации в сайте oriC (желтый круг) и продвижение репликационной вилки (ter, серая полоса). Инициация репликации запускается АТФ-связанной формой белка-инициатора DnaA. DnaA активируется через кислые фосфолипиды в составе клеточной мембраны через хромосомные сайты DARS1 и DARS2; дезактивация белка происходит через сайт datA (механизм регуляторной инактивации). DnaA имеет высокое сродство к сайтам титрования на бактериальной хромосоме (серые круги). B. Модель титрования. Инициирующая ДНК (красные круги) может связываться как с oriC (желтый круг), так и с хромосомными сайтами титрования. C. Модель переключения инициатора. В модели Lipid-DatA активация и инактивация DnaA регулируется кислыми фосфолипидами и сайтом datA, соответственно. В расширенной модели Lipid-DatA-DARS1/2-RIDA в качестве активирующих сайтов выступают DARS1 и DARS2. Основным механизмом снижения активности DnaA становится регуляторная инактивация.
1
Традиционно модель «титрования» и модель «накопления инициатора» рассматривались как независимые механизмы. С использованием математических методов ученым удалось найти «место пересечения» двух важнейших регуляторных процессов. Данная работа – отличный пример перехода от практики, т.е. экспериментальных лабораторных методов, к теории – созданию математических моделей, позволяющих описать, а, может быть, и предсказать явления, которые мы все еще не можем изучить в лабораторных условиях. Следующая задача – доказать результаты экспериментально.
Литература
1. Berger M., Wolde P.R.t. Robust replication initiation from coupled homeostatic mechanisms. Nature Communications, 2022.
2. Лихошвай В.А., Хлебодарова Т.М. Согласование темпов роста объема клетки и репликации ДНК: математическая модель. Математическая биология и биоинформатика, 2013.
3. Amir A. Cell size regulation in bacteria. Physical Review Letters, 2014.
4. Hansen F.G. et al. The initiator titration model: computer simulation of chromosome and minichromosome control. Research in Microbiology, 1991.
5. Hansen F.G., Atlung T. The DnaA Tale. Frontiers in Microbiology, 2018.




.jpg)