Медиатека
Автостопом по методам работы с РНК
Оглавление:
•
Исследование транскриптома и секвенирование РНК
•
РНК-интерференция
•
in vitro
транскрипция
•
Визуализация РНК
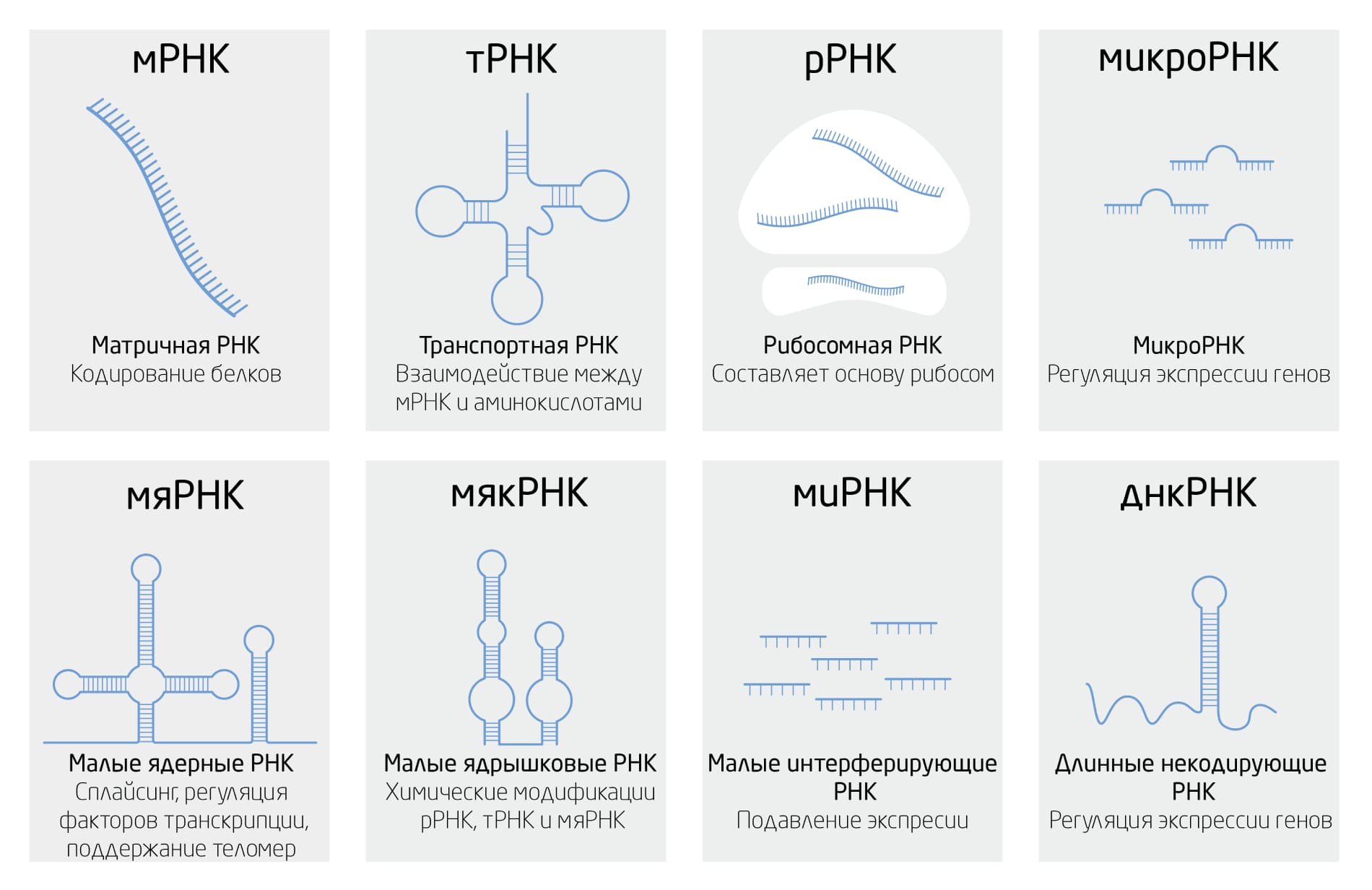
Все молекулы нужны, все молекулы важны в контексте функционирования и жизнедеятельности клеток. Особый класс молекул представляют РНК (Рис. 1). Они разнообразны по структуре, длине и функциям: матричные РНК (мРНК) выступают посредниками в передаче генетической информации, транспортные РНК (тРНК) переносят аминокислоты, рибосомные РНК организуют пространственную структуру рибосом, многочисленный подкласс малых РНК длиной менее 200 нуклеотидов выполняет важные регуляторные и сигнальные функции и т.д. Работе с разными типами РНК предъявляются разные требования. Основные – соблюдение аккуратности и чистоты реактивов и оборудования. Молекулы РНК более нестабильные, чем молекулы ДНК, и легко разрушаются при несоблюдении правил работы и хранения.
В продолжение цикла статей про биологические молекулы мы рассмотрим ряд классических и современных методов работы с РНК (преимущественно с мРНК).
Рис. 1. Виды РНК.
Исследование транскриптома и секвенирование РНК
Транскрипция генов в комплементарные молекулы РНК (транскриптом) определяет клеточную идентичность и регулирует биологические процессы внутри клетки. Ранние исследования экспрессии генов на уровне транскрипции основывались на малопроизводительных методах, таких как нозерн-блоттинг и количественная ПЦР, которые ограничивались измерением одиночных транскриптов. За последние 20 лет были разработаны методы, объединенные термином транскриптомика, которые позволяют проводить полногеномную количественную оценку экспрессии генов. 1
Первые транскриптомные исследования были проведены с использованием технологии микрочипов на основе гибридизации 2, которая обеспечивает высокую производительность при относительно низкой стоимости. В отличие от методов, основанных на гибридизации, подходы на основе секвенирования были разработаны для анализа транскриптома путем непосредственного определения нуклеотидной последовательности транскрипта. Первоначально в исследованиях генной экспрессии применяли создание библиотек маркерных экспрессированных последовательностей (EST) посредством секвенирования комплементарной ДНК (кДНК) по Сэнгеру 3, но этот подход отличается низкой производительностью и не совсем подходит для количественного определения транскриптов. Чтобы преодолеть эти ограничения, были разработанные методы, основанные на тегах (метках), такие как серийный анализ экспрессии генов (SAGE) 4 и анализ экспрессии генов по кэпу (CAGE) 5. Эти методы обеспечивают более высокую производительность и более точную количественную оценку уровней экспрессии, но при этом нечувствительны к изменению уровней экспрессии разных сплайсированных изоформ, а также не могут быть использованы для открытия новых генов.
Секвенирование РНК (RNA-Seq) – самый современный и эффективный метод оценки уровня экспрессии генов, который использует возможности методов высокопроизводительного секвенирования для получения наиболее полного представления о транскриптоме клетки 1, 6. По сравнению с предыдущими методами RNA-Seq обеспечивает гораздо более широкий охват и лучшее разрешение для анализа динамической природы транскриптома. Помимо количественной оценки экспрессии генов, данные, полученные с помощью RNA-Seq, облегчают открытие новых транскриптов, идентификацию альтернативно сплайсированных генов и обнаружение аллель-специфичной экспрессии. Наряду с секвенированием транскриптов полиаденилированных матричных РНК (мРНК) RNA-Seq можно применять для исследования различных популяций РНК, включая тотальную РНК, пре-мРНК и некодирующие РНК (нкРНК), такие как микроРНК и длинные нкРНК. Вершина современного транскриптомного анализа – секвенирование транскриптов единичных клеток (scRNA-seq) 7.
Рабочий процесс по секвенированию РНК состоит из выделения тотальной клеточной РНК, преобразования ее в кДНК методом обратной транскрипции, подготовки библиотеки и последующего секвенирования на платформе NGS (Рис. 2).
Рис. 2. Методология секвенирования РНК. Сначала из выбранного биологического материала (клеток, тканей) выделяют тотальную РНК. Затем нужную фракцию молекул РНК выделяют с использованием специального протокола (например, для обогащения библиотеки полиаденилированными транскриптами или для удаления рибосомной РНК). Далее РНК конвертируют в кДНК с помощью обратной транскрипции и к концам фрагментов кДНК лигируют адаптеры для секвенирования.(например, с использованием готовых наборов для подготовки направленных РНК-библиотек). После амплификации методом ПЦР библиотека готова к секвенированию. 1
Транслятом – это группа мРНК, которая непосредственно транслируется в белки на рибосомах в данный момент времени. Для анализа этой группы используется рибосомный профайлинг. Этот метод также основан на глубоком секвенировании РНК, но при этом фрагменты РНК экстрагируются напрямую из рибосом, которые отделяют от других компонентов клетки путем центрифугирования в сахарозном градиенте. Обычно одновременно с транслятомом секвенируется и траснкриптом. Сравнив их, можно сделать вывод, какие РНК (мРНК) транслируются в клетке, насколько эффективно и как регулируются эти процессы. 8
РНК-интерференция
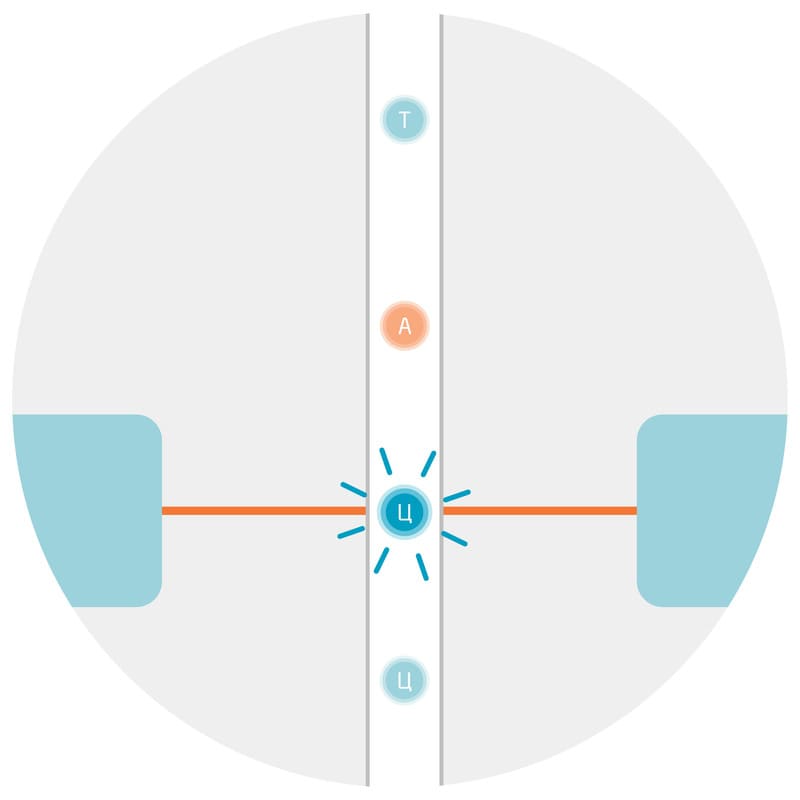
Посредством РНК регулируются разные клеточные процессы. РНК-интерференция – пример пост-транскрипционной регуляции, в результате которой элиминируются целевые мРНК и таким образом подавляется экспрессия гена (нокдаун гена). В основе РНК-интерференции лежит каскад реакций, который начинается с узнавания и расщепления двухцепочечной РНК до коротких молекул (малых интерферирующих РНК, миРНК/siRNA) особой эндонуклеазой Dicer (Рис. 3). Затем миРНК в комплексе с эндонуклеазой Argonaute и другими белками (RISC, RNA-induced silencing complex) находят молекулы РНК, комплементарные миРНК, и инактивируют их 9. Весь процесс уже достаточно хорошо изучен и взят на вооружение учеными для изучения функций генов. Для нокдауна целевого гена достаточно внести в клетки комплементарную искусственно синтезированную двухцепочечную РНК. В отличии от нокаута (полного выключения), нокдаун генов с помощью РНК-интерференции носит временный и контролируемый характер. Благодаря этому данный подход можно использовать в терапии онкологических, сердечно-сосудистых и нейродегенеративных заболеваний 10.
Рис. 3. Схема РНК-интерференции.
in vitro транскрипция
РНК можно синтезировать искусственными методами 11 и внедрить в клетки. Таким образом можно, например, экспрессировать нужный белок. Иногда этот способ предпочтительнее доставки плазмидной ДНК, так как нет необходимости в стадии транскрипции и при экспорте мРНК из ядра. При попадании искусственной мРНК в цитоплазму она начинает сразу транслироваться, если, конечно, мРНК соответствует клеточным «требованиям». В случае эукариотических мРНК важно наличие кэп-структуры на 5`-конце мРНК, поли-А хвоста на 3`-конце, отсутствие интронов в кодирующей части, а также наличие стартового кодона в «хорошем» контексте (контекст Козак), иначе рибосома просто не распознает начальную точку трансляции и вхолостую проскользнет вдоль мРНК. Для синтеза РНК in vitro обычно используют реакцию транскрипции с использованием фермента РНК-полимеразы. Она строит цепочку РНК на основе матрицы ДНК, а дополнительные реакции позволяют достроить поли-А хвост и присоединить кэп. Доставить готовую синтезированную и очищенную РНК в клетки можно несколькими способами: химической трансфекцией клеток, электропорацией и микроинъекцией. Но важно не «перегружать» клетки экзогенной РНК, так как это может послужить большим стрессом для клеток. Есть вероятность, что вместо того, чтобы активно экспрессировать мРНК, клетка запустит антистрессовый ответ и ингибирует трансляцию.
Визуализация РНК
Мониторинг РНК позволяет исследователям визуализировать молекулы РНК в клетках и тканях, предоставляя важную пространственно-временную информацию о динамике и функциях РНК. Такие методы, как флуоресцентная гибридизация in situ (FISH) и молекулярные маяки, основаны на комплементарных олигонуклеотидах для мечения и визуализации эндогенных транскриптов. Другие методы подразумевают создание искусственных химерных транскриптов, которые связываются с флуоресцентно меченными белками (например, белками оболочки из бактериофагов MS2 и λN) и позволяют отслеживать такие комплексы в живых клетках в режиме реального времени. В других подходах эндогенные РНК распознаются комплементарными РНК в комплексе с некаталитическими белками Cas. Каждый метод имеет свои сильные стороны и ограничения (сведены в Таблице 1), которые необходимо учитывать при планировании эксперимента. 12
Таблица 1. Обзор методов мониторинга РНК. 13
Разнообразие методов работы с РНК велико, как и многообразие самих РНК. Длительная эволюция этих молекул, которые гораздо более древние, чем ДНК и белки, позволила им захватить самые разнообразные биологические ниши в устройстве и жизнедеятельности клеток. Учёные до сих пор обнаруживают новые типы и классы РНК. Например, недавно открытые глико-РНК, функция которых пока неизвестна 13. Это значит, что арсенал методов работы с РНК также будет активно расширяться.
Литература
1. Kukurba K.R., Montgomery S.B. RNA Sequencing and Analysis. Cold Spring Harbor Protocols, 2015.
2. Schulze A., Downward J. Navigating gene expression using microarrays — a technology review. Nature Cell Biology, 2001.
3. Adams M.D. et al. Complementary DNA sequencing: expressed sequence tags and human genome project. Science, 1991.
4. Velculescu V.E., Zhang L., Vogelstein B., Kinzler K.W. Serial analysis of gene expression. Science, 1995.
5. Shiraki T. et al. Cap analysis gene expression for high-throughput analysis of transcriptional starting point and identification of promoter usage. Proceedings of the National Academy of Sciences, 2003.
6. Wang Z., Gerstein M., Snyder M. RNA-Seq: a revolutionary tool for transcriptomics. Nature Reviews Genetics, 2009.
7. Chen G., Ning B., Shi T. Single-Cell RNA-Seq Technologies and Related Computational Data Analysis. Frontiers in Genetics, 2019.
8. Ingolia N.T., Hussmann J.A., Weissman J.S. Ribosome Profiling: Global Views of Translation. Cold Spring Harbor Perspectives in Biology, 2019.
9. Sashital DG, Doudna JA. Structural insights into RNA interference. Current Opinion in Structural Biology, 2010.
10. Pecot C.V. et al. RNA interference in the clinic: challenges and future directions. Nature Reviews Cancer, 2011.
11. Beckert B., Masquida B. Synthesis of RNA by in vitro transcription. Methods in Molecular Biology, 2011.
12. George L., Indig F.E., Abdelmohsen K., Gorospe M. Intracellular RNA-tracking methods. Open Biology, 2018.
13. Flynn R.A. et al. Small RNAs are modified with N-glycans and displayed on the surface of living cells. Cell, 2021.




.jpg)



.jpg)
.jpg)