Медиатека
QIAseq – от образца к открытию
Секвенирование нового поколения (Next Generation Sequencing, NGS) – это группа методов, которые позволяют быстро и точно установить нуклеотидную последовательность нуклеиновых кислот. Данный метод имеет широкое применение, позволяя изучать геномы самых разных организмов. Это приводит к значительным достижениям и новым открытиям в различных исследовательских областях.
Чтобы усовершенствовать исследования на основе NGS и помочь ученым в достижении своих целей, QIAGEN разработал линейку продуктов QIAseq. Линейка QIAseq предлагает специализированные решения1 возможных проблем на любом этапе секвенирования – от подготовки образцов до анализа:
- Таргетные панели QIAseq представляют собой экономически эффективный способ таргетного обогащения, не усложняя при этом процессы анализа и интерпретации данных.
- Продукты для подготовки библиотеки QIAseq позволяют провести секвенирование всего генома или транскриптома и избежать ошибок, связанных с ПЦР-дубликатами.
- QIAseq предлагает оптимизированный рабочий процесс без ПЦР для изучения полных геномов или транскриптомов одиночных клеток.
- QIAGEN Bioinformatics сочетает в себе лучшее в своем классе программное обеспечение для анализа и интерпретации данных NGS с доступом к уникальной базе знаний, курируемой экспертами.
Уникальные молекулярные индексы UMI
Уникальные молекулярные идентификаторы/индексы (Unique Molecular Indices, UMI) – это молекулярные маркеры, обеспечивающие коррекцию ошибок секвенирования и повышенную точность получаемых данных. Они представляют собой2 короткие олигонуклеотидные последовательности, используемые для уникальной маркировки каждого фрагмента ДНК в NGS-библиотеке до амплификации.
Многие коммерчески доступные методы подготовки библиотек используют полимеразную цепную реакцию, которая зачастую является источником возможных ошибок, связанных, например, с ПЦР-дубликатами. Это значительно ограничивает вероятность обнаружения истинных редких вариантов. Таргетные панели QIAseq преодолевают эти проблемы за счет использования UMI.
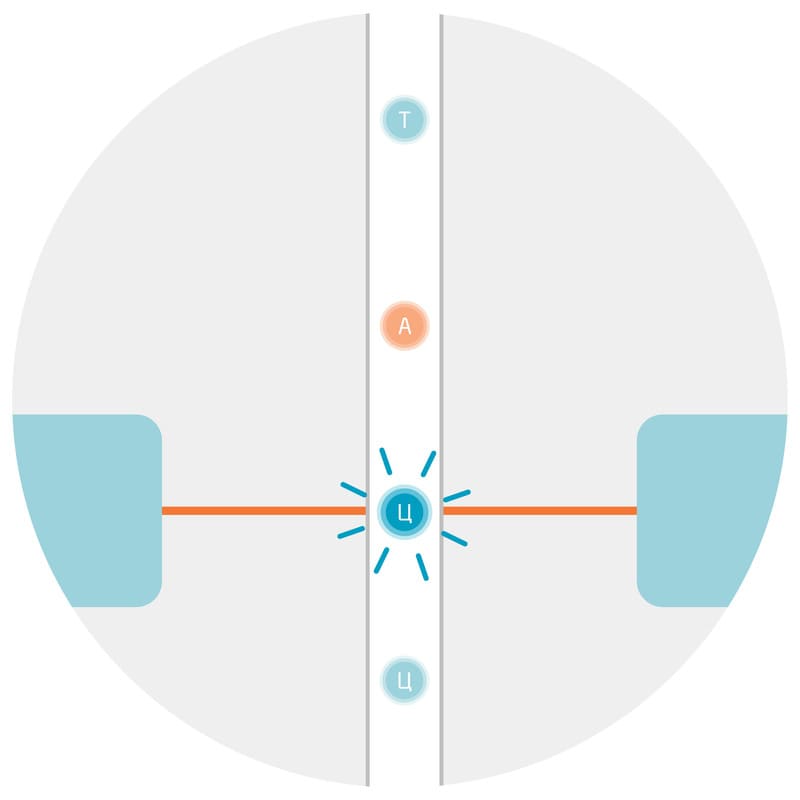
UMI играют роль уникальных маркеров3 фрагментов ДНК библиотеки. Таким образом, даже при неравномерной амплификации можно получить равномерное покрытие всего генома путем подсчета количества UMI, а не общего количества чтений. Чтения, имеющие разные UMI, представляют собой разные исходные молекулы, в то время как чтения последовательностей, имеющие одинаковые UMI, являются копиями4 одной исходной молекулы.
Рис.1. Принцип устранения ошибок и отклонений при подсчете чтений с помощью молекулярных индексов UMI.
Также можно отличить вариантные аллели, присутствующие в исходном образце, от ложноположительных результатов, возникших вследствие ошибок, допущенных во время подготовки библиотеки и секвенирования.
Рис.2. Принцип3 обнаружения истинных вариантных аллелей с помощью уникальных молекулярных индексов UMI.
Уникальные двойные индексы UDI
При использовании уникальных двойных индексов (UDI) все молекулы ДНК в образце помечаются двумя уникальными индексами, что обеспечивает более надежное обнаружение редких вариантов и более точное демультиплексирование при параллельном секвенировании нескольких библиотек в одном цикле.
Уникальные двойные индексы позволяют увеличить5 количество образцов, секвенируемых за цикл, до 96, и в связи с этим снизить затраты на образец по сравнению с другими стратегиями индексирования.
Рис 3. Сравнение5 уникальных и комбинаторных двойных индексов.
Помимо уникальных двойных индексов возможно использование комбинаторных двойных индексов. При подготовке библиотек рекомендуется использовать уникальные двойные индексы, так как они помогают выполнять демультиплексирование с повышенной точностью.
Рис 4. Уменьшение ошибок6 вследствие контаминации с помощью уникальной двойной индексации.
К преимуществам уникального двойного индексирования относятся:
- Более высокая эффективность мультиплексирования и демультиплексирования.
- Снижение затрат на образец за счет включения большего количества индексов в цикл секвенирования по сравнению с двойными комбинаторными или одиночными индексами.
- Снижение ошибок, связанных с неверным присвоением индекса при подготовке образцов, секвенировании и анализе.
Удлинение праймера с одного конца (Single primer extension)
Чтобы преодолеть проблемы и возможные ошибки, связанные с ПЦР-амплификацией в процессе NGS, помимо уникальных молекулярных индексов UMI используется технология7 single primer extension (SPE), которая обеспечивает гибкость в дизайне и высокоспецифичную селекцию нужных геномных участков.
После удаления неиспользованных адаптеров проводится определенное количество циклов ПЦР с использованием адаптерного праймера Illumina и пула одиночных праймеров, каждый из которых несет ген-специфичную последовательность и универсальную 5'-последовательность. При этом нарабатывается один и тот же целевой локус из разных ДНК-матриц. После этого проводятся дополнительные циклы ПЦР с использованием универсальных праймеров для присоединения адаптеров Illumina и последующей амплификации библиотеки.
Рис 5. Принцип метода SPE.
По сравнению с существующими подходами таргетного обогащения, метод SPE основан на лигировании адаптера к одному концу фрагмента двухцепочечной ДНК, что имеет гораздо более высокую эффективность, чем лигирование адаптеров к обоим концам фрагмента двухцепочечной ДНК. Как следствие, больше молекул ДНК доступно для дальнейшего этапа обогащения в ходе ПЦР.
Заключение
В настоящее время идет бурное развитие методов исследования молекулярных структур живых организмов. Появление методов секвенирования нового поколения позволяет геномике развиваться быстрее и использовать сложные технологии и многоэтапные протоколы, что приводит к ошибкам и недостоверным результатам в процессе проведения исследований.
В связи с этим QIAGEN предлагает новые решения. Линейка QIAseq позволяет получать данные секвенирования высокого качества, значительно облегчая работу исследователей и гарантируя коррекцию возможных ошибок на всех этапах.
Литература
1. Introducing QIAseqTM. QIAGEN, 2017.
2. Kavaklioglu T. How to solve major sequencing pitfalls with Unique Molecular Identifiers (UMIs). GenomeScan, 2019.
3. QIAseq Targeted DNA Panel Handbook. QIAGEN, 2021.
4. How can unique molecular identifiers (UMIs) help to reduce quantitative biases? ecSeq Bioinformatics, 2017.
5. Understanding unique dual indexes (UDI) and associated library prep kits. Illumina, 2021.
6. Packer H. A solution for sample crosstalk and index hopping in multiplexed NGS. Integrated DNA Technologies, 2017.
7. Samara R. QIAseq and Single Primer Extension Chemistry. QIAGEN, 2018.




.jpg)