Медиатека
Просто о сложном: технология секвенирования cPAS
Разберемся, в чем секрет высокой точности прочтений на секвенаторах BGI.
Технология секвенирования, используемая в приборах DNBSEQ производства BGI/MGI, является модернизированной технологией американской компании Complete Genomics. Это один из методов секвенирования путем синтеза, но он имеет ряд особенностей и преимуществ, позволяющих улучшить качество получаемых данных.
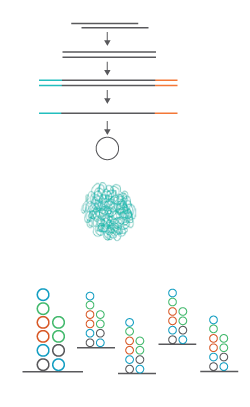
Технологический процесс можно условно разделить на 3 этапа:

|
|
|
 |
|
|
 |
|
1. Подготовка библиотек
Первый этап пробоподготовки — фрагментация ДНК до фрагментов длиной 200-500 п.н. Ее можно проводить с помощью ультразвука или ферментов. После репарации концов ДНК к фрагментам лигируются специальные адаптеры. Данные адаптеры комплементарны двум участкам олигонуклеотида, который добавляется в реакцию на следующем этапе. Добавление такого олигонуклеотида делает возможным циркуляризацию исходного фрагмента ДНК, а сам олигонуклеотид служит затравкой при последующей амплификации по типу катящегося кольца – амплификации с множественным вытеснением цепи (Rolling circle amplification, RCA) (рис.1).

|

|
|

|
|

|

|
| Добавление олигонуклеотида | Циркуляризация | Достраивание цепи ДНК | Амплификация по типу «катящегося кольца» |
Рис. 1. Амплификация по типу катящегося кольца.
Данный тип клональной амплификации хорош тем, что матрицей в каждом цикле служит исходный фрагмент ДНК и накопление продукта происходит линейно. За счет этого исключается аккумулирование ошибок ДНК-полимеразы в ходе ПЦР, а также снижается количественное искажение библиотек, что значительно повышает точность детекции однонуклеотидых вариантов и инделов.
2. Формирование наношариков ДНК
Амплификацию проводят до тех пор, пока количество копий исходной матрицы не достигнет 300-500. Благодаря запатентованной структуре олигонуклеотидов и адаптеров длинная цепь ДНК укладывается в компактную структуру, так называемый наношарик (DNA nanoball, DNB) (рис.2)
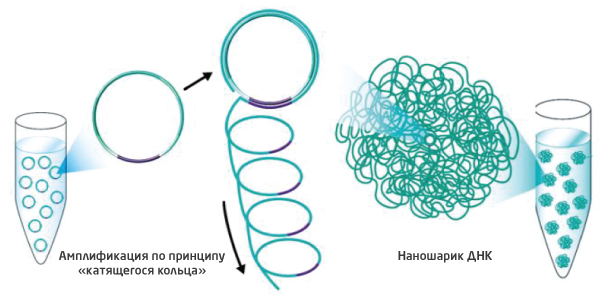
Рис. 2. Формирование наношарика необходимо для многократного увеличения интенсивности флуоресцентного сигнала от одной точки при последующем секвенировании.
Наглядное видео технологии формирования наношариков:
Полученную библиотеку фрагментов ДНК в виде наношариков наносят на специальную проточную ячейку (рис.3)

Рис. 3. Нанесение наношариков ДНК на регуляризированную ячейку.
В зависимости от модели секвенатора проточные ячейки (Flow Cell, FC) (рис.4) могут состоять из 1, 2 или 4 дорожек. Каждая дорожка несет заряженные участки, к которым за счет электростатических сил присоединяются наношарики. Расстояние между заряженными участками рассчитаны таким образом, что к каждому участку присоединяется один наношарик. Проточная ячейка содержит несколько миллиардов заряженных участков. На практике наношариками заполняются около 90% из них.
Проточная ячейка легко заполняется вручную, нет необходимости использовать автоматическую станцию пробоподготовки**.

A.

B.
Рис. 4. Проточные ячейки. А. Проточная ячейка для секвенатора DNBSEQ-G50 содержит 1 дорожку. B. Проточная ячейка типа FCL (Flow Cell Large) с 4 дорожками для секвенатора DNBSEQ-G400.
3. Секвенирование по принципу cPAS (рис.5)
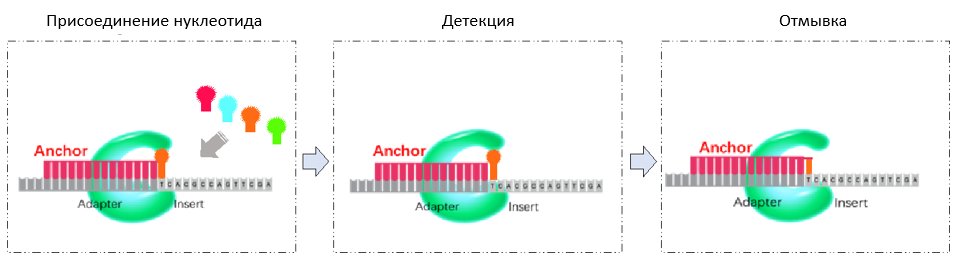
Рис. 5. Принцип секвенирования cPAS.
cPAS (Combinatorial Probe-Anchor Synthesis) — модифицированный вариант метода секвенирования cPAL (Combinatorial Probe-Anchor Ligation), разработанный компанией Complete Genomics. В нем сочетаются преимущества запатентованной технологии формирования DNB и секвенирования синтезом: отсутствие клонального накопления ошибок, низкое количественное искажение библиотек, признанную высокую точность.
На первом этапе к наношарикам добавляется праймер для секвенирования (Ancor — якорная последовательность, якорь). Он присоединяется к адаптерной последовательности ДНК-наношарика. Далее в реакцию добавляется 4 вида нуклеотидов, каждый из которых мечен индивидуальным* флуоресцентным красителем. Нуклеотид присоединяется к комплементарно синтезируемой последовательности ДНК (Insert на Рис. 5). После его присоединения лишние неспецифичные нуклеотиды удаляются из реакции. Флуоресцентная метка на присоединившемся нуклеотиде возбуждается лазером и сигнал детектируется CCD-камерой. После детекции флуоресцентная метка отщепляется, и проточная ячейка промывается.
При повторении циклов присоединения нуклеотида и детекции сигнала происходит секвенирование исходной цепи ДНК (одноконцевое прочтение, SE).
В зависимости от необходимой длины прочтений можно проводить от 50 до 400 циклов! (режимы SE50, SE100, SE400).
Если необходимы парноконцевые (PE) прочтения, проводится синтез обратной цепи ДНК. К наношарику добавляются нуклеотиды и высокоточная высокопроцессивная ДНК-полимераза phi29, инициирующая амплификацию с множественным вытеснением цепи (Multiple displacement amplification, MDA) (рис.6). За счет этого значительно сокращается время синтеза второй цепи. Свойства полимеразы phi29 позволяют проводить достраивание цепей ДНК на исходной и всех дочерних цепях одновременно.

Рис. 6. Принцип синтеза комплементарной цепи для секвенирования обратной цепи.
После MDA-реакции к наношарикам добавляются обратные праймеры и проводится секвенирование обратной цепи ДНК.
В случае, если образцы баркодированы, после секвенирования обратной цепи ДНК к наношарикам добавляются праймеры для баркодов и проводится их секвенирование.
Полученные в итоге данные имеют стандартный формат FASTQ, что позволяет анализировать их привычным биоинформатическим ПО.
Технологии, используемые в приборах BGI/MGI, — создание DNB, секвенирование по типу cPAS, использование регуляризованной ячейки, — уникальны по своей точности и позволяют не только получать данные высокого качества, но также экономить время и бюджет.
Основные приложения секвенаторов BGI:
- Геномное и экзомное секвенирование.
- Секвенирование de novo.
- Метагеномика.
- Таргетное секвенирование.
- Репродуктивная генетика.
- Секвенирование транскриптома.
На данный момент на российском рынке представлено два геномных секвенатора MGI – DNBSEQ-G50 и DNBSEQ-G400 с производительностью 150 Гб и 1440 Гб за запуск соответственно.
Для получения дополнительной информации по технологии, приборам, расходным материалам и реагентам для пробоподготовки можете написать на электронную почту mail@helicon.ru.
*Количество флуоресцентных красителей на пробах зависит от типа реагентов. В секвенаторе DNBSEQ-G400 используется 4-х цветная химия (каждый из 4 олигонуклеотидов мечен индивидуальным флуорофором), в DNBSEQ-G50 – 2-х цветная (2 из 4 олигонуклеотидов мечены индивидуальными флуорофорами, 3-й мечен сразу двумя красителями, 4-й не несет метки). Кроме того, для секвенатора DNBSEQ-G400 доступен принципиально новый тип реагентов CoolMPS. Эта технология использует флуоресцентно-меченные антитела к нуклеотидам каждого типа и позволяет получать данные еще более высокого качества по показателю Q30!
16 августа 2023 года получено регистрационное удостоверение Росздравнадзора 2023/20825 на полногеномные секвенаторы HELICON® G50 и HELICON® G400! |




.jpg)